图 GLORI 2.0和3.0实现痕量样本全转录检测m6A修饰
在国家自然科学基金项目(批准号:22425071)等资助下,北京大学伊成器团队联合中科院遗传与发育生物研究所王秀杰团队,于2025年5月5日在《自然・方法》 (Nature Methods)期刊上发表了题为“Mild and ultrafast GLORI enables absolute quantification of m6A methylome from low-input samples”的研究论文,论文链接为https://www.nature.com/articles/s41592-025-02680-9。该研究攻克了表观转录组学领域的关键技术瓶颈,建立了适用于痕量样本的m6A绝对定量检测新技术。
N6-甲基腺嘌呤(m6A)是真核生物mRNA中最普遍的核心化学修饰之一,该修饰通过调控可变剪接、RNA转运及翻译稳定性等分子机制,在基因表达调控中发挥"分子开关"作用。研究团队前期建立了基于特异性脱氨反应的化学测序技术GLORI;然而,现有条件仍存在RNA降解、起始样本需求量大等局限性,限制了其在低输入样本中的广泛应用。
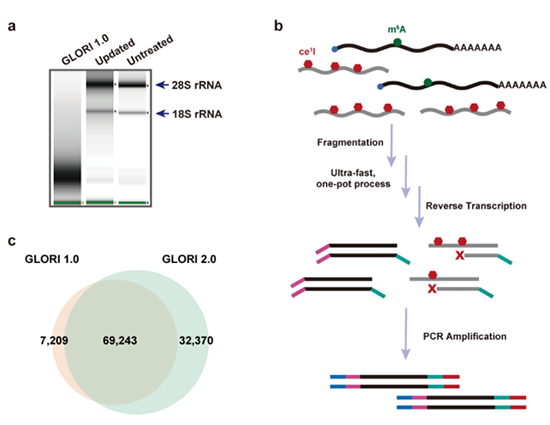
针对上述问题,该研究团队首先深入剖析了GLORI介导的脱氨机制,在此基础上优化反应条件,成功构建了新一代m6A检测方法— GLORI 2.0。该方法采用温和、快速且操作便捷的一锅式的反应流程,显著提升了RNA样品的完整性,大幅降低了对RNA起始量的需求(图a)。为进一步提升技术灵敏度,研究团队创新性地设计了一种反转录沉默载体RNA(RT-silencing carrier RNA),可在文库构建过程中自动被移除,从而避免对后续测序结果造成干扰。将该策略整合进GLORI 2.0体系后,研究人员进一步开发出GLORI 3.0技术,实现了从几百至几千个细胞中精准绘制m6A修饰图谱(图b)。实验验证表明,相较于原始GLORI方法,GLORI 2.0在多个关键指标上表现出显著优势:首先,其RNA起始量需求显著降低;其次,能够有效识别更多位于低丰度mRNA上的m6A修饰位点(图c)。而GLORI 3.0在保持类似定量性能的同时,更适合处理超低起始量RNA样本。为评估GLORI 3.0的实际应用潜力,研究团队将其应用于小鼠海马体突触和细胞质组分的m6A修饰分析,结果发现Slitrk3、Bdnf等突触相关基因具有较高的m6A修饰水平,提示该技术在低起始量样本中的应用可行性。
经过系统优化后的GLORI技术显著提升了m6A修饰检测的灵敏度与适用性,能够高效应对少量乃至痕量RNA样本的挑战。这一进展对于解析复杂组织亚细胞成分、异质性稀有细胞类型以及稀缺临床样本的表观转录组学研究具有重要意义。